Oceani predstavljajo enega od najslabše raziskanih ekosistemov. Če se omejimo samo na bakterije, ocenjujejo, da v morjih živi okoli 2 milijona bakterijskih vrst. Ta številka je precej v navzkrižju s številom vseh bakterijskih vrst, pri katerih poznamo vsaj malo dednine. V podatkovni zbirki GenBank, kjer hranijo DNA zaporedja, je opisanih le 30.000 bakterijskih vrst. Za ta razkorak je pomembno dejstvo, da se bakterijske vrste, ki živijo v ekstremnih okoljih, zelo težko goji v laboratorijskih razmerah. V laboratoriju je namreč težko zagotoviti specifične snovi, ki jih bakterije potrebujejo za rast in so posledica odnosov med različnimi vrstami, ki potekajo v naravi. Okoljski vzorci so zato po pravilu osiromašeni in ne predstavljajo prave slike o strukturi in dinamiki bakterijskih populacij. Pred kratkim je Craig Venter s svojo skupino v reviji Science objavil članek, ki morda ponuja rešitev. Za analizo bakterijskih vrst v Sargaškem morju so uporabili isto tehnologijo, kot pri določanju nukleotidnega zaporedja človeškega genoma.
Venterjevi dosežki v genomiki
 Craig Venter velja za enega od najbolj pomembnih raziskovalcev v genomiki, vedi ki se ukvarja z analizo dednine v živih organizmih. S svojimi eksperimentalnimi pristopi je pomembno zaznamoval razvoj genomike in vplival na skoraj vsa področja biologije in medicine v zadnjih petnajstih letih. Še posebej pomembni so naslednji trije mejniki:
Craig Venter velja za enega od najbolj pomembnih raziskovalcev v genomiki, vedi ki se ukvarja z analizo dednine v živih organizmih. S svojimi eksperimentalnimi pristopi je pomembno zaznamoval razvoj genomike in vplival na skoraj vsa področja biologije in medicine v zadnjih petnajstih letih. Še posebej pomembni so naslednji trije mejniki:
- Analiza izraženih oznak zaporedij (ang. »expressed sequence tags«)- Izražene oznake zaporedij dobimo po posebnem postopku iz obveščevalne RNA. Ta iz jedra, kjer so zapisani geni na kromosomih, prenaša informacijo o zgradbi proteinov v citoplazmo, kjer poteka njihova izgradnja. Vsaka oznaka izraženih zaporedij predstavlja en gen, ki je bil v celici v tistem trenutku aktiven. Če nam torej uspe določiti zaporedje večine obveščevalnih RNA v celici, dobimo prerez aktivnosti genoma. S to metodo je možno primerjati aktivnosti genov pri različnih tipih celic, npr. zdravi in rakavi. Poiščemo lahko tiste gene, ki so pri bolezenskih stanjih bolj ali manj aktivni in torej predstavljajo morebitne tarče za zdravila. Zato se je metoda kmalu prijela tudi v farmacevtskih firmah in ostalih akademskih laboratorijih. Danes večina zaporedij v GenBank predstavljajo ravno ta zaporedja, t.i. EST kloni. V zadnji izdaji jih je malo več kot 24 milijonov. Venter je to uporabo te metode prvič opisal v reviji Science 1991. leta. (Complementary DNA sequencing: expressed sequence tags and human genome project.Adams, M.D., J.M. Kelley, J.D. Gocayne, M. Dubnick, M.H. Polymeropoulos, H. Xiao, C. R. Merril, A. Wu, B. Olde, R.F. Moreno, A.R. Kerlavage, W.R. McCombie, and J.C. Venter. 1991.Science 252: 1651–1656).
 Človeški genom– Venter se je v tekmo za človeški genom vključil sorazmerno pozno, ob napovedih javnega konzorcija, da bo projekt končan šele okoli leta 2003. Projekta se je lotil s spremenjeno metodo in velikanskim finančnim vložkom. Povezal se je s proizvajalci aparatov za določevanje nukleotidnega zaporedja in računalniškim podjetjem Compaq.
Človeški genom– Venter se je v tekmo za človeški genom vključil sorazmerno pozno, ob napovedih javnega konzorcija, da bo projekt končan šele okoli leta 2003. Projekta se je lotil s spremenjeno metodo in velikanskim finančnim vložkom. Povezal se je s proizvajalci aparatov za določevanje nukleotidnega zaporedja in računalniškim podjetjem Compaq.  Leta 1998 so ustanovili podjetje Celera Genomics (slika levo), kjer so za potrebe človeškega genoma sestavili drugi najbolj zmogljiv računalnik v ZDA. Človeški genom so tako določali v pravih »tovarnah«, kjer so uporabljali 300 najbolj zmogljivih aparatov za določanje nukleotidnega zaporedja in 200 Compaqovih alfa postaj za analizo dobljenih podatkov.
Leta 1998 so ustanovili podjetje Celera Genomics (slika levo), kjer so za potrebe človeškega genoma sestavili drugi najbolj zmogljiv računalnik v ZDA. Človeški genom so tako določali v pravih »tovarnah«, kjer so uporabljali 300 najbolj zmogljivih aparatov za določanje nukleotidnega zaporedja in 200 Compaqovih alfa postaj za analizo dobljenih podatkov.
Metodo določanja zaporedja genoma z uporabo naključnih kratkih fragmentov je Venter razvil že leta 1995 in z njo so sploh določili prvi kompleten genom bakterije Haemophilus influenzae, nato pa so jo v okviru Celere testirali še na višjem organizmu. Leta 1999 so določili zaporedje vinske mušice, ki je v biologiji pomemben modelni sistem predvsem pri študiju razvoja. Takoj nato so začeli z določevanjem zaporedja človeškega genoma in aprila 2000 so v izjavi za tisk obvestili svetovno javnost, da so končali z grobim osnutkom človeškega genoma. Na objavo v znanstvenih revijah smo čakali še pol leta, ko sta februarja 2001 Celera in javni konzorcij objavila izsledke v revijah Science (The sequence of the human genome. Venter, J.C. et al. 2001. Science 291:1304-1351) in Nature (Initial sequencing and analysis of the human genome. The Genome International Sequencing Consortium. 2001. Nature 409, 860-921).
(The sequence of the human genome. Venter, J.C. et al. 2001. Science 291:1304-1351) in Nature (Initial sequencing and analysis of the human genome. The Genome International Sequencing Consortium. 2001. Nature 409, 860-921).
- Okoljska genomika– Že ob zaključku projekta človeški genom se je Venter začel ozirati v prihodnost. Leta 2002 je zapustil mesto direktorja Celere. Predlagal je, da bi svojo tehnologijo določevanja nukleotidnega zaporedja prenesel na področje ekologije. Z dovolj zmogljivimi računalniki lahko preiščeš ogromno količino zaporedij v GenBank in poveš nekaj o okoljskem vzorcu, predvsem pa odkriješ nove gene in genome. Tukaj odpade zahtevno in zamudno gojenje mikroorganizmov. Venter si je za testiranje metod izbral vzorec Sargaškega morja. Izsledke pa so pred kratkim objavili v reviji Science (Environmental genome shotgun sequencing of the Sargasso Sea. Venter, J.C. et al. 2004.Science 304:66-74.).
Analiza bakterijskih vrst v Sargaškem morju
 O uporabi genomskih metod pri opisu okoljskih vzorcev je Venter govoril že ob sklenitvi projekta človeški genom leta 2001. Pri svojih projektih se zanaša na moč izjemno zmogljivih računalnikov. Za določevanje zaporedja 3 milijard nukleotidov, ki sestavljajo človeški genom, je uporabil metodo naključnih kratkih fragmentov (ang. »whole genom shutgun sequencing«). V tej metodi se pri enem branju naenkrat naključno določi 500-1000 nukleotidov dolgo zaporedja iz genoma. Zaporedja v naslednji fazi računalnik primerja med seboj in jih zloži v končno verzijo. Pri tem si izdatno pomagajo z DNA zaporedji, ki so shranjena v podatkovnih zbirkah, predvsem v GenBank, kjer je zaenkrat shranjenih 32.5 milijona zaporedij. Venter se tudi pri okoljskih vzorcih zanaša na moč računalnikov in podatke iz GenBank. Bistvo njegove ideje je v tem, da iz okoljskega vzorca izolira celotno DNA, ne da bi pri tem izoliral posamezne organizme. Nato določa nukleotidna zaporedja ne glede na izvor, jih primerja s shranjenimi v GenBank in iz pojavljanja zaporedij nekaj pove o raznolikosti vzorca. Če odkrijejo zaporedja, ki v GenBank še niso shranjena, pa po vsej verjetnosti pripadajo neki novi, še neopisani vrsti. (Slika desno: Craig Venter na krovu ladje Sorcerer II.)
O uporabi genomskih metod pri opisu okoljskih vzorcev je Venter govoril že ob sklenitvi projekta človeški genom leta 2001. Pri svojih projektih se zanaša na moč izjemno zmogljivih računalnikov. Za določevanje zaporedja 3 milijard nukleotidov, ki sestavljajo človeški genom, je uporabil metodo naključnih kratkih fragmentov (ang. »whole genom shutgun sequencing«). V tej metodi se pri enem branju naenkrat naključno določi 500-1000 nukleotidov dolgo zaporedja iz genoma. Zaporedja v naslednji fazi računalnik primerja med seboj in jih zloži v končno verzijo. Pri tem si izdatno pomagajo z DNA zaporedji, ki so shranjena v podatkovnih zbirkah, predvsem v GenBank, kjer je zaenkrat shranjenih 32.5 milijona zaporedij. Venter se tudi pri okoljskih vzorcih zanaša na moč računalnikov in podatke iz GenBank. Bistvo njegove ideje je v tem, da iz okoljskega vzorca izolira celotno DNA, ne da bi pri tem izoliral posamezne organizme. Nato določa nukleotidna zaporedja ne glede na izvor, jih primerja s shranjenimi v GenBank in iz pojavljanja zaporedij nekaj pove o raznolikosti vzorca. Če odkrijejo zaporedja, ki v GenBank še niso shranjena, pa po vsej verjetnosti pripadajo neki novi, še neopisani vrsti. (Slika desno: Craig Venter na krovu ladje Sorcerer II.)
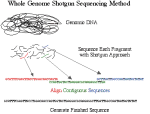 Določanje nukleotidnega zaporedja genomov preko naključnih kratkih fragmentov
Določanje nukleotidnega zaporedja genomov preko naključnih kratkih fragmentov
Pri tej metodi (ang. »whole genom shutgun sequencing«) genomsko DNA, ki je v celicah v obliki kromosomov, mehansko razbijemo na manjše fragmente, dolge okoli 2000 nukleotidov. Fragmentom določimo nukleotidno zaporedje in jih v zadnji fazi zložimo skupaj v končano zaporedje preko delov, ki se prekrivajo. Na sliki je to zaporedje šestih nukleotidov na začetku in koncu fragmentov. Tak pristop zahteva predvsem zelo močno računalniško podporo.
 Venter si je izbral Sargaško morje za pilotno študijo, da bi preveril svojo idejo. V njem živi malo različnih vrst, ker je revno s hranili. Poleg tega tam že dolgo deluje raziskovalna postaja in na voljo je veliko dodatnih fizikalnih podatkov. Skozi posebej zgrajen filtrirni sistem (slika desno) so prefiltrirali ogromno količino morske vode in na filtrih zadržali večinoma bakterije in viruse.
Venter si je izbral Sargaško morje za pilotno študijo, da bi preveril svojo idejo. V njem živi malo različnih vrst, ker je revno s hranili. Poleg tega tam že dolgo deluje raziskovalna postaja in na voljo je veliko dodatnih fizikalnih podatkov. Skozi posebej zgrajen filtrirni sistem (slika desno) so prefiltrirali ogromno količino morske vode in na filtrih zadržali večinoma bakterije in viruse.
V laboratoriju so iz organizmov na filtrih izolirali DNA in določili nukleotidna zaporedja 1.66 milijonu zaporedij, ki so predstavljala več kot 1.2 milijona genov. Z računalniško analizo so uspeli sestaviti 12 kompletnih bakterijskih genomov. Iz podatkov ocenjujejo, da je v vzorcu Sargaškega morja vsaj 1800 bakterijskih vrst in preko 70.000 povsem novih genov. Od teh je pet vrst zastopanih v večjem številu. Kljub temu, da je bil glavni namen študije opis bakterijskih populacij je metoda več kot primerna za analizo druge vrste DNA iz okoljskih vzorcev, npr. plazmidov ali pa evkariontskih mikroogranizmov (npr. alg, gliv). Prav tako je uporabna za druge ekosisteme, npr. za mikroorganizme v zemlji. Podatki Sargaškega morja so shranjeni v GenBank v novem odseku vzorcev okoljske genomike.
 Venter pa se tukaj še ni ustavil. Trenutno poteka odprava Sorcerer II, ki jo financirajo Venterjeva znanstvena fundacija, TV kanal Discovery in Ministrstvo ZDA za energijo. Odprave, ki je bila inspirirana z odpravami Beagle in Challenger, bo na poti okoli sveta zbrala kopenske in predvsem vzorce morske vode vseh svetovnih morij in nekaterih kopenskih ekosistemov. Ladja na vsakih 200 milj prefiltrira 200 litrov morske vode z globine 2 m. Filtrirni sistem je zasnovan tako, da lahko ločijo organizme po velikosti in zamrznjene na filtrih shranijo za kasnejšo analizo. Na dvoletno pot okoli sveta se je Sorcerer II odpravila avgusta 2003 iz Halifaxa v Kanadi. Trenutno plujejo ob obalah Avstralije. (Potek odprave Sorcerer II na sliki desno.)
Venter pa se tukaj še ni ustavil. Trenutno poteka odprava Sorcerer II, ki jo financirajo Venterjeva znanstvena fundacija, TV kanal Discovery in Ministrstvo ZDA za energijo. Odprave, ki je bila inspirirana z odpravami Beagle in Challenger, bo na poti okoli sveta zbrala kopenske in predvsem vzorce morske vode vseh svetovnih morij in nekaterih kopenskih ekosistemov. Ladja na vsakih 200 milj prefiltrira 200 litrov morske vode z globine 2 m. Filtrirni sistem je zasnovan tako, da lahko ločijo organizme po velikosti in zamrznjene na filtrih shranijo za kasnejšo analizo. Na dvoletno pot okoli sveta se je Sorcerer II odpravila avgusta 2003 iz Halifaxa v Kanadi. Trenutno plujejo ob obalah Avstralije. (Potek odprave Sorcerer II na sliki desno.)
Venter ocenjuje, da bodo po končani analizi z vseh mest, kjer nameravajo zbrati vzorce, določili preko nekaj deset milijonov novih genov in več stotisoč vrst novih bakterij. Pristop bo gotovo popolnoma spremenil razumevanje mikroorganizmov, zato se primerjava odprave z ladjo Beagle in drugimi odpravami v 19. stoletju ponuja kar sama. Za sorazmerno majhen denar, bomo dobili neprecenljive podatke o mikrobni raznolikosti v oceanih in vplivih onesnaževanja nanje.
Povezave
- GenBank– podatkovna zbirka, ki vsebuje DNA zaporedja. GenBank je ena od večjih podatkovnih zbirk v biologiji, saj trenutno vsebuje 32.5 milijona zaporedij.
- Mikrobna diverziteta– članek o pričetkih okoljske genomike na Nacionalnem centru za biotehnološko informacijo v ZDA.
- SorcererII– ekspedicija, katere namen je zbrati okoljske vzorce vseh večjih morij in z metodo naključnega določanja nukleotidnega zaporedja analizirati genetski material.
- Bioinformatika – iskanje igle v kopici sena
(Gregor Anderluh)

















